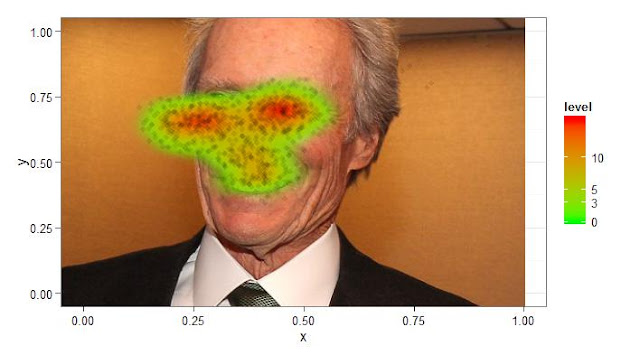
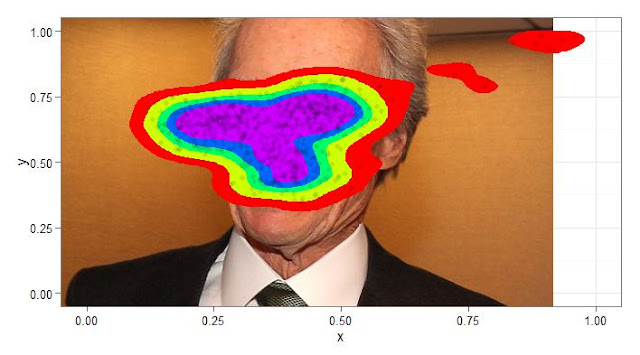
Esta semana quería seguir con el tema del Eyetracker, pero
las respuestas que he encontrado me han llevado a nuevas preguntas y aún está
demasiado flojo el tema como para dar una respuesta a esta duda. Así que lo dejamos
( esperemos) para la semana que viene. Os diré que casi he conseguido evitar
los diagramas de densidad y dibujar frecuencias como algunos de vosotros pedíais.
Lo único es que me gustaría estructurarlo, porque la verdad es que he
encontrado unas 5 soluciones que espero que os gusten. Así, volvernos a
una vieja entrada que es va de como limpiar datos que nuca está de más.
Hace ya tiempo que me vi obligado a tratar una serie de BD portuguesas para hacer un analisis de su liga (Está todavía muy en primeros pasos, pero me gustaría en el futuro mostraros algo). Esto causaba multiples problemas en el trato de caracteres sobre todo al guardar en .RData y cambiar de sistema operativo. Ahora os ofrezco una solución con un poquito de scrapeo de webs fáciles como Wikipedia gracias a librería rvest. El script es el siguiente y lo podeis ejecutar sin problemas sobre vuestra consola R.
Un saludo a todos y muchas gracias por las visitas de esta semana que han sido una gran recompensa para el trabajo que he hecho.
Hace ya tiempo que me vi obligado a tratar una serie de BD portuguesas para hacer un analisis de su liga (Está todavía muy en primeros pasos, pero me gustaría en el futuro mostraros algo). Esto causaba multiples problemas en el trato de caracteres sobre todo al guardar en .RData y cambiar de sistema operativo. Ahora os ofrezco una solución con un poquito de scrapeo de webs fáciles como Wikipedia gracias a librería rvest. El script es el siguiente y lo podeis ejecutar sin problemas sobre vuestra consola R.
Un saludo a todos y muchas gracias por las visitas de esta semana que han sido una gran recompensa para el trabajo que he hecho.